- TOP
- よくわかるLAMP法
- RT-LAMP法
RT-LAMP法
RT-LAMP法とは
RT-LAMP(Reverse Transcription - Loop - mediated Isothermal Amplification)法は、LAMP法を応用し、標的遺伝子であるRNAからcDNAを合成して増幅・検出する方法です。
サンプルとなるRNAに対して、DNAを標的遺伝子とする場合と同様の試薬(プライマー、鎖置換型DNA合成酵素、基質等)に逆転写酵素を追加して混合することによって、一定温度(60~65℃)で増幅から検出までの工程を1ステップで行うことができます。
プライマー設計
Target RNAの塩基配列におけるU(ウラシル)をT(チミン)に置き換えた配列に対して、LAMP法と同様に6つの領域を用いて4種類のプライマーを設計します。
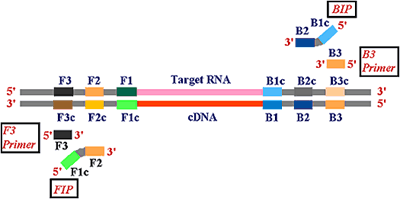
原理
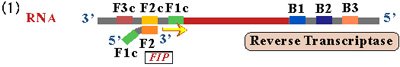
原理図 ; RT-LAMP(+鎖)
- 逆転写反応(RT)ステップ
STEP1
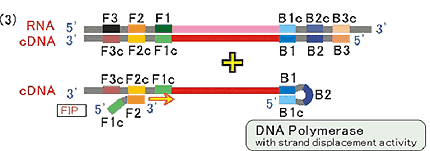
ウイルスRNA分離試薬を用いてヒト由来のRNAを抽出し、サンプル溶液を調製します。サンプル溶液と反応液とを混合し、60~65℃でインキュベートすることにより、下図(1)に示すように対象のRNAにBIPがアニールし、逆転写酵素により相補的なcDNAを合成します。

STEP2
BIPの外側にB3 Primerがアニールし、逆転写酵素の働きにより、先にBIPから伸長合成されたcDNA鎖を剥がしながら、新たな相補的なcDNAを合成します。

STEP3
(2)のステップにより、BIPから伸長合成した後に1本鎖DNA状態となったcDNAに対してFIPがアニールします。
- 起点構造ができるまでのステップ
STEP4
上記の逆転写反応ステップの(3)から、鎖置換型DNAポリメラーゼの働きにより、FIPのF2領域の3’末端を起点として、鋳型となるcDNAと相補的なDNA鎖が合成されます。

STEP5
FIPの外側にF3 Primerがアニールし、その3 ’末端を起点として、先に合成されているFIPからのDNA鎖を剥がしながらDNAが伸長していきます。
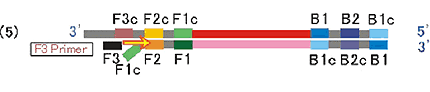
STEP6
F3 Primerから合成されたDNA鎖と鋳型DNAが2本鎖となります。
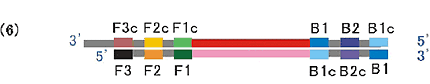
STEP7
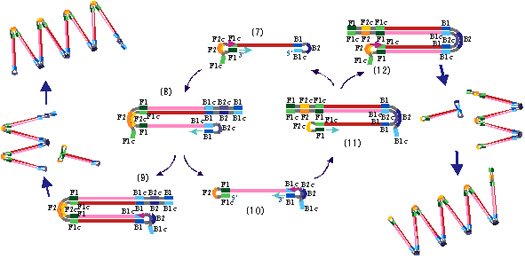
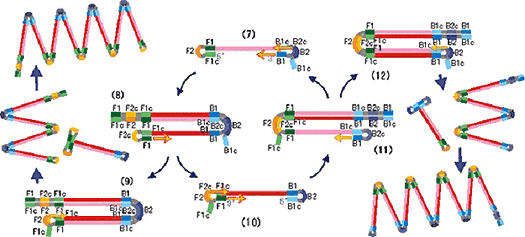
(5)の過程ではがされたFIPから合成されたDNA鎖は両端に相補的な配列を持つため自己アニールし、ループを形成してダンベル型の構造となります。この(7)がLAMP法における増幅サイクルの起点構造となります。
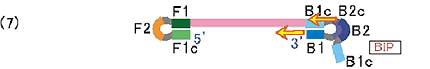
- LAMP法増幅サイクルステップ
STEP8
まず(7)の構造で、3’末端のB1領域を起点として自己を鋳型としたDNA合成が伸長し、この時、5’末端側のループは剥がされて伸びます。更に、3’末端側のループのB2c領域は1本鎖であるためBIPがアニールすることができ、そのB2領域の3’末端を起点として、先に合成されているB1領域からのDNA鎖を剥がしながらDNA合成が伸長していきます。
STEP9
次に(8)の構造において、BIPから伸長合成されたDNA鎖によって剥がされて1本鎖となったB1領域から伸長したDNA鎖は、その3’末端側に相補的な領域を持つためループを形成します。このループのF1領域の3’末端から、1本鎖となった自己を鋳型としてDNA合成が始まります。そして、そのDNA鎖が2本鎖部分となっているBIPからのDNA鎖を剥がしながら伸長し、(9)の構造となります。
STEP10
上記過程によって、BIPから合成されたDNA鎖は1本鎖となり、その両端にそれぞれ相補的な領域、F1、F1cおよびB1、B1cを持っているため自己アニールしてループを形成し、(10)の構造となります。この(10)の構造は、先ほどの(7)の構造と全く相補的な構造となります。
STEP11
(10)の構造では、(7)の場合と同様にF1領域の3’末端を起点として自己を鋳型としたDNA合成が行われ、さらに1本鎖となっているF2c領域にFIPがアニールしてF1領域からのDNA鎖を剥がしながらDNA合成が行われます。それにより、ちょうど(7)、(8)、(10)の過程と同様に(10)、(11)の過程を経て再び(7)の構造ができます。
STEP12
また、(9)あるいは(12)の構造において、1本鎖となっているF2c(あるいはB2c)領域にFIP(あるいはBIP)がアニールし、2本鎖部分を剥がしながらDNA鎖が合成されます。そして、これらの過程の結果、同一鎖上に互いに相補的な配列を繰り返す構造の増幅産物がいろいろなサイズでできてきます。
原理図 ; RT-LAMP(−鎖)
- 逆転写反応(RT)ステップ
STEP1
ウイルスRNA分離試薬を用いてヒト由来のRNAを抽出し、サンプル溶液を調製します。サンプル溶液と反応液とを混合し、60~65℃でインキュベートすることにより、下図(1)に示すように対象のRNAにFIPがアニールし、逆転写酵素により相補的なcDNAを合成します。
STEP2
FIPの外側にF3 Primerがアニールし、逆転写酵素の働きにより、先にFIPから伸長合成されたcDNA鎖を剥がしながら、新たな相補的なcDNAを合成します。

STEP3
(2)のステップにより、FIPから伸長合成した後に1本鎖DNA状態となったcDNAに対してBIPがアニールします。
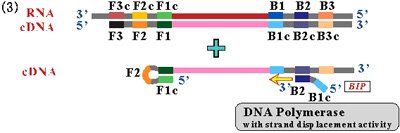
- 起点構造ができるまでのステップ
STEP4
上記の逆転写反応ステップの(3)から、鎖置換型DNAポリメラーゼの働きにより、BIPのB2領域の3’末端を起点として、鋳型となるcDNAと相補的なDNA鎖が合成されます。

STEP5
BIPの外側にB3 Primerがアニールし、その3’末端を起点として、先に合成されているBIPからのDNA鎖を剥がしながらDNAが伸長していきます。

STEP6
B3 Primerから合成されたDNA鎖と鋳型DNAが2本鎖となります。

STEP7
(5)の過程ではがされたBIPから合成されたDNA鎖は両端に相補的な配列を持つため自己アニールし、ループを形成してダンベル型の構造となります。この(7)がLAMP法における増幅サイクルの起点構造となります。

- LAMP法増幅サイクルステップ
STEP8
まず(7)の構造で、3’末端のF1領域を起点として自己を鋳型としたDNA合成が伸長し、この時、5’末端側のループは剥がされて伸びます。更に、3’末端側のループのF2c領域は1本鎖であるためFIPがアニールすることができ、そのF2領域の3’末端を起点として、先に合成されているF1領域からのDNA鎖を剥がしながらDNA合成が伸長していきます。
STEP9
次に(8)の構造において、FIPから伸長合成されたDNA鎖によって剥がされて1本鎖となったF1領域から伸長したDNA鎖は、その3’末端側に相補的な領域を持つためループを形成します。このループのB1領域の3’末端から、1本鎖となった自己を鋳型としてDNA合成が始まります。そして、そのDNA鎖が2本鎖部分となっているFIPからのDNA鎖を剥がしながら伸長し、(9)の構造となります。
STEP10
上記の過程によって、FIPから合成されたDNA鎖は1本鎖となり、その両端にそれぞれ相補的な領域、F1、F1cおよびB1、B1cを持っているため自己アニールしてループを形成し、(10)の構造となります。この(10)の構造は、先ほどの(7)の構造と全く相補的な構造となります。
STEP11
(10)の構造では、(7)の場合と同様にB1領域の3’末端を起点として自己を鋳型としたDNA合成が行われ、さらに1本鎖となっているB2c領域にBIPがアニールしてB1領域からのDNA鎖を剥がしながらDNA合成が行われます。それにより、ちょうど(7)、(8)、(10)の過程と同様に(10)、(11)の過程を経て再び(7)の構造ができます。
STEP12
また、(9)あるいは(12)の構造において、1本鎖となっているB2c(あるいはF2c)領域にBIP(あるいはFIP)がアニールし、2本鎖部分を剥がしながらDNA鎖が合成されます。そして、これらの過程の結果、同一鎖上に互いに相補的な配列を繰り返す構造の増幅産物がいろいろなサイズでできてきます。